SALSA® MLPA® probemix P036 SUBTELOMERES MIX 1
Katalogové číslo # P036-025R, P036-50R, P036-100R
Název produktu: Probemix P036 Subtelomeres Mix 1
Šarže: E2-0413
25, 50 nebo 100 reakcí.
Přepravní podmínky: Dodává se v suchém ledu nebo s chladicími prvky.
Skladujte při teplotách mezi -25 °C a -15 °C.
Doba použití: Nejméně až do dubna 2018 po otevření při skladování v doporučených podmínkách.
Tento produkt lze zmrazit / rozmrazit alespoň 20krát.
Použití
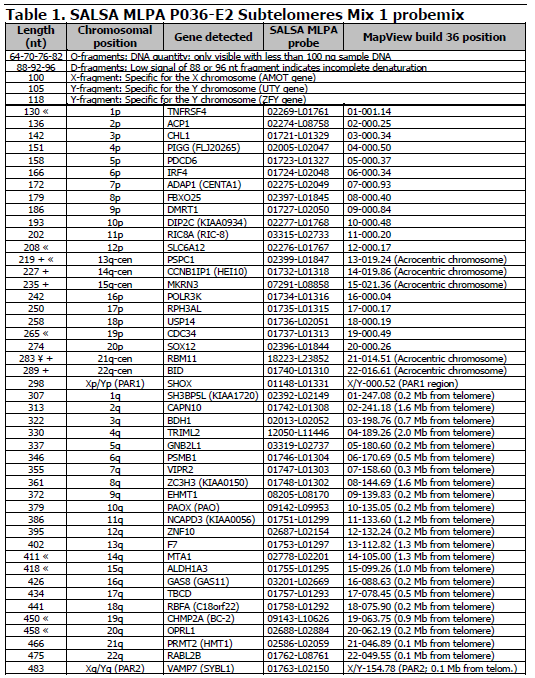
Tento produkt byl vyvinut pro stanovení počtu kopií DNA jedné sekvence v každé ze 41 lidských subtelomerických oblastí. Žádné sondy nejsou přítomny pro p-raménka pěti akrocentrických chromozomů. U těchto pěti chromozomů je přítomna navíc sonda, která detekuje sekvenci na q raménku v blízkosti centromery. Lokalizace sond jsou uvedeny v Tabulce 1 v Popisu produktu. Tento kit by měl být použit v kombinaci s kitem SALSA MLPA EK1 (100 reakcí) nebo EK5 (500 reakcí), jak je popsáno v MLPA obecném protokolu.
Test kvality 1
Dostatečná vzdálenost mezi píky sond, absence zvláštních píků, úplná hybridizace každé sondy, testováno na sekvenátorech Applied Biosystems 3130 a Beckman GeXP. SPLNĚNO
Test kvality 2
Variabilita každé jednotlivé sondy <0,10 při testu na 23 různých DNA vzorcích zdravých jedinců, izolace různými způsoby. SPLNĚNO
Test kvality 3
Variabilita každé jednotlivé sondy testována na stejném vzorku DNA při různých experimentálních podmínkách, které splňují kritéria pro specifickou reakci. Podmínky byly testovány napodobováním pipetovacích chyb; odpařováním; odchýlením teploty v termocykleru; přítomností solí, které mají vliv na denaturaci DNA, nebo přítomností nečistot ve vzorku DNA. SPLNĚNO
Test kvality 4
Reakce vzorku bez DNA má za výsledek pouze 5 hlavních píků kratších než 120 nt: 4 Q - gragmenty o délce 64-70-76-82 nt a nepoužitou část 19nt fluorescenčních PCR primerů. Žádné nespecifické píky delší než 120 nt a výška píků více než 25 % mediánu 4 Q - fragmentů. (Poznámka: u píků pod 25% hranicí se neočekává, že by měly vliv na MLPA reakci při použití dostatečného množství vzorku DNA.) SPLNĚNO
Evropa *:
FOR IN VITRO DIAGNOSTIC (IVD) USE. THIS PRODUCT IS CE MARKED.
Pro in vitro diagnostické (IVD) účely. Tento produkt je označen značkou CE.
Mimo Evropu: FOR RESEARCH USE ONLY (RUO). Pouze pro výzkumné účely (RUO).
* zahrnuje členské státy EU, kandidátní státy pro vstup do Evropské unie a členy Evropského sdružení volného obchodu (European Free Trade Association - EFTA). Výrobek je určen pro RUO ve všech ostatních zemích v rámci Evropy.
Žádná ze složek kitu není odvozena z lidí, zvířat nebo patogenních bakterií. Na základě koncentrací žádná ze složek v současné době není nebezpečná, jak je definováno v Hazard Communication Standard. MSDS list není nutný pro tyto produkty: žádný z přípravků neobsahuje nebezpečné látky (podle směrnice 67/548/EEC a změny) v koncentracích, které vyžadují přiložení MSDS listu (podle směrnice 1999/45/EEC, 2001/58/EEC). Pokud dojde k rozlití, omyjte čistou vodou a dodržujte příslušné místní postupy.
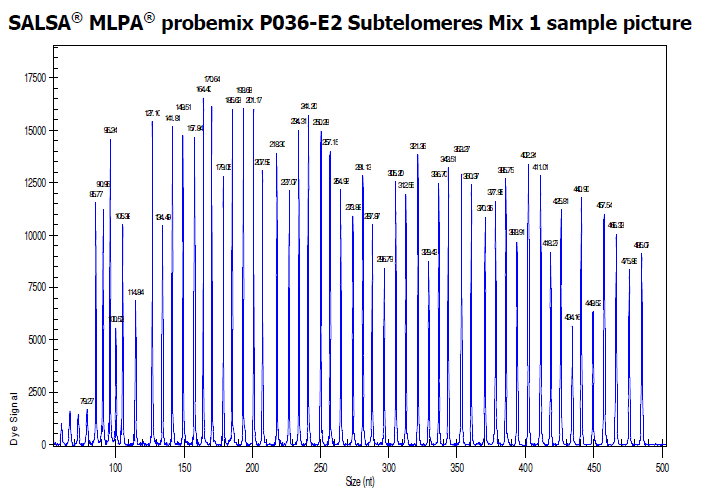
Obrázek 1. Profil píků z kapilární elektroforézy vzorku lidské mužské kontrolní DNA (přibližně 50 ng) analyzované kitem SALSA MLPA® Probemix P036-E2 Subtelomeres Mix 1 (šarže E2-0413).
Popis produktu
SALSA® MLPA® probemix P036-E2 SUBTELOMERES MIX 1
Použijte s obecným protokolem pro MLPA.
Verze E2.
Katalogová čísla:
- P036-025R: SALSA® MLPA® probemix P036 Subtelomeres Mix 1, 25 reakcí.
- P036-050R: SALSA® MLPA® probemix P036 Subtelomeres Mix 1, 50 reakcí.
- P036-100R: SALSA® MLPA® probemix P036 Subtelomeres Mix 1, 100 reakcí.
Kity by měly být použity v kombinaci se SALSA MLPA reagent kit EK1 (100 reakcí) nebo EK5 (500 reakcí). MLPA kity jsou k dispozici s různými fluorescenčními barvivy: EK1-FAM, EK5-FAM (pro Applied Biosystems sekvenátory) a EK1-Cy5.0, EK5-Cy5.0 (pro Beckman sekvenátory).
Použití: Tento kit SALSA MLPA probemix P036 Subtelomeres Mix 1 je in vitro diagnostický test pro detekci delecí a / nebo duplikací v subtelomerických oblastech lidské DNA z periferní krve, bukální sliznice a (ne)kultivovaných amniocytů nebo choriových klků, které mohou být potenciální příčinou zpožděného vývoje, dysmorfických znaků, jiných vrozených vad a / nebo ztráty těhotenství. Zjištěné abnormality by měly být vždy potvrzeny příslušným MLPA navazujícím kitem nebo jinou metodou.
Klinické pozadí: Aberantní počet kopií subtelomerických oblastí, např. kvůli nebalancované
translokaci, je častou příčinou vývojového opoždění a vrozených vad. Tento kit P036-E2
Subtelomeres Mix 1 probemix pro MLPA poskytuje rychlejší způsob (s vyšší výkonností a menšími ekonomickými náklady) identifikace jedinců se změnou počtu kopií v jedné nebo více subtelomerických oblastech než microarray analýza a FISH. Pomocí MLPA nelze nalézt balancované translokace a metoda má nižší detekční rychlost než microarray analýza. Míra detekce silně závisí na souboru testovaných pacientů. Příkladem je 5,9% (Ahn et al, 2007) při testování 455 pacientů nebo 3,9% (Stegmann et al, 2008) při testování kohorty pacientů, ve které normální analýza G-pruhováním nezjistila žádné abnormality. Všimněte si, že výsledky Ahn et al. zahrnují některé vzorky obsahující genetickou aberaci, která byla zděděna od zdravého rodiče a která tudíž může představovat klinicky bezvýznamný polymorfismus. Viz níže několik publikací o kitu P036 Subtelomeres Mix 1.
MLPA metoda: Princip MLPA metody je popsán v obecném protokolu.
Obsah kitu P036-E2: Kit SALSA MLPA probemix P036 Subtelomeres Mix 1 obsahuje 47 MLPA sond s amplifikačními produkty mezi 118 a 483 nt: 2 sondy pro každý chromozom a 1 sonda pro jedinečnou část chromozomu Y. 41 sond je umístěno v subtelomerických oblastech. Nejsou přítomny sondy pro subtelomerické oblasti 5 akrocentrických chromozomů (13, 14, 15, 21, 22). Pro tyto chromozomy je zde navíc sonda pro detekci q raménka v blízkosti centromery. Subtelomerické sondy pro X a Y chromozom jsou identické, protože detekují sekvence v pseudoautozomální oblastech (PAR1 a PAR2), které jsou totožné u chromozomu X a Y. Více informací je k dispozici v Tabulce 1 tohoto dokumentu.
Tento kit obsahuje devět kontrol pro kvalitu fragmentů, které generují amplifikační produkty mezi 64 a 105 nt: čtyři DNA fragmenty pro kvantitu (Q-fragmenty), tři DNA fragmenty pro denaturaci (D-fragmenty), jeden chromozom X a jeden Y chromozom specifický fragment (Tabulka 1). Více informací o tom, jak interpretovat výsledky u těchto kontrolních fragmentů lze nalézt v MLPA obecném protokolu.
Charakteristika výkonu metody: Přesná charakteristika výkonu nemůže být snadno určena z důvodu obtíží při získávání dostatečného počtu vzorků DNA, které obsahují delece nebo duplikace pro všechny sondy. Ahn et al (2007) stanovili v jejich laboratoři 95% interval spolehlivosti pro pozitivní a negativní predikci s přesnosti 0,951-0,996 a 0,9996 - 1.
Výkon metody může být ohrožen mimo jiné: nečistotami ve vzorku DNA, neúplnou DNA denaturací, použitím nedostatečného nebo naopak velkého množství DNA, použitím nevhodných referenčních vzorků, problémy s kapilární elektroforézou nebo špatným postupem při normalizaci dat. MLPA obecný protokol obsahuje technické pokyny a informace o vyhodnocení dat /normalizaci. Interní validace metody MLPA ve Vaší laboratoři je nezbytná a měla by vykazovat standardní odchylku u každé sondy 0,10 nebo nižší při testování vzorků DNA od zdravých jedinců.
Limitace metody:
- Pomocí MLPA nelze zjistit žádné změny, které leží mimo cílovou sekvenci sond a nelze zjistit většinu inverzí a balancovaných translokací. U mnoha subtelomerických oblastí byly popsány patogenní intersticiální delece a duplikace, které mohou být snadno nedetekovány jedinou P036 sondou pro danou subtelomerickou oblast. I když pomocí MLPA nebyly zjištěny žádné odchylky, stále je možnost, že existují biologické změny v této chromozomální oblasti, ale nebyly detekovány.
- Při použití (ne)kultivovaných amniocytů, kontaminace vzorku mateřskou DNA může vést k nesprávným závěrům.
- Při použití choriových klků, rozdíly v chromozomálních profilech DNA u choriových klků plodu byly popsány v důsledku maternální kontaminace, postzygotické nondisjunkce, postzygotické isochromozomové formace, mozaiky a komplikaci při odběru vzorků DNA od dvojčat (Van den Berg et al, 2006).
- Ne všechny subtelomerické abnormality detekované pomocí MLPA jsou patogenní. V případě pozitivních výsledků, důrazně doporučujeme, aby byli také otestováni rodiče. Pro každou subtelomerickou oblast je determinován počet kopií pouze jedné malé (70 nt) sekvence. Tyto sekvence byly vybrány na základě jejich blízkosti k teloméře a jsou obvykle umístěny v genech, které nebyly přímo zapojeny do opožděného vývoje nebo vrozených vad. U mnoho chromozomů není známo, jaká minimální velikost je nutná pro subtelomerickou deleci / duplikaci, která má za následek opožděný vývoj nebo vrozené vady. A konečně, četnost benigních změn v počtu kopií (CNV) je vyšší v subtelomerických oblastech (http://dgv.tcag.ca/dgv/app/home).
- Sekvenční změny (např. SNP, bodové mutace, malé indels) v cílové sekvenci sondy mohou způsobit falešně pozitivní výsledky. Mutace / SNP (přinejmenším až 20 nt od vazebního místa sondy) může snižovat signál sondy tím, že brání ligaci oligonukleotidové sondy nebo destabilizuje navázání sondy na DNA.
- Kompletní sekvence detekováné sondami jsou k dispozici na mlpa.com.
Požadavky na vzorky: DNA izolovaná z periferní krve, bukální sliznice a (ne) kultivovaných amniocytů nebo choriových klků, zbavená nečistot. Pro více informací, viz Obecný MLPA protokol a mlpa.com – část věnovaná přípravě DNA vzorků.
Referenční vzorky: Vzorky referenční DNA by měly být odvozeny ze stejné tkáně a měly by být izolovány stejným způsobem jako vzorky pacientů. Referenční vzorky by měly být odvozeny od nebříbuzné osoby bez opožděného vývoje nebo vrozených vad. Více informací o použití referenčních vzorků lze nalézt v Obecném MLPA protokolu.
Pozitivní kontroly: Vzorek syntetické pozitivní DNA s duplikací (p/n SD025 DNA; RUO) je k dispozici. SD025 DNA vykazuje duplikaci 2-4 sond při použití v kombinaci s kity P036, P069, P070, P245, P064, P290. SD025 DNA je směs lidské ženské DNA, která byla opatrně titrována linearizovaným plasmidem, který obsahuje různé cílové sekvence sondy.
Analýza dat: Pro analýzu dat musí být software Coffalyser.Net použit v kombinaci s vhodným MLPA produktový listem specifickým pro danou šarži (volně ke stažení na mlpa.com). Metoda analýzy používána pro P036-E2 je „bloková“ normalizace: všechny sondy, s výjimkou 118 nt chromozom Y-specifické sondy, se používají jako referenční sondy. Normalizace výsledků by měla být provedena v rámci jednoho experimentu. Pro více podrobné informace o kontrole kvality MLPA a analýze dat, viz manuál Coffalyser.Net.
Validace výsledků: Změny v počtu kopií detekované pouze jedinou sondou vždy vyžadují potvrzení jinou metodou. Zřejmá delece detekována jedinou sondou může být způsobena např. mutací / polymorfismem, který zabraňuje ligaci nebo destabilizuje vazbu mezi oligonukleotidovou sondou a vzorkem DNA. Sekvenční analýza může stanovit, zda jsou přítomny v cílové sekvenci sondy mutace a polymorfismy. Nález heterozygotní mutace nebo polymorfismu znamená, že dvě různé alely jsou přítomny ve vzorku DNA, a proto byl získán falešně pozitivní výsledek pomocí MLPA.
Vzhledem k vysoké pravděpodobnosti falešně pozitivních / negativních výsledků, je doporučeno používat oba kity MLPA probemix P036 a MLPA probemix P069 nebo P070 pro počáteční testování každého vzorku DNA. Všechny P069 a P070 sondy se liší od P036 sond. Změny v počtu kopií detekované P036 a P069 nebo P070 mohou být bez klinického významu. V případě pozitivních výsledků, proto důrazně doporučujeme, aby byli také testováni rodiče. V případě, že P036 a P069/P070 dávají protichůdné výsledky, jsou zapotřebí další studie. Pro potvrzení a vymezení rozsahu subtelomerické delece/duplikace, MLPA navazující kity obsahující 6-12 sond pro konkrétní subtelomerickou oblast jsou k dispozici. Potvrzení může být provedeno také pomocí FISH, microarray nebo sekvenováním genomu s nízkým pokrytím. Potvrzení výsledků může být obtížné: delece / duplikace mohou být intersticiální a nemusí zasahovat až k telomeře. P036 a P070 sondy mohou detekovat sekvence, které jsou umístěny poměrně daleko od sebe nebo daleko od komerčně dostupných FISH sond.
Interpretace výsledků: Očekávaný počet alelických kopií pro 118 nt chromozom Y specifickou sondu je 0 (žena) nebo 1 (muž). Očekávaný počet kopií u všech ostatních sond, včetně sond pro pseudoautozomální oblasti pohlavních chromozomů, je 2 (normál), 1 (heterozygotní delece) nebo 3 (heterozygotní duplikace). Opožděný vývoj nebo jiné abnormality v důsledku subtelomerických delecí nebo duplikací jsou ve většině případů autozomálně dominantní onemocnění. Přestavba jednoho chromozomu může být patogenní. Delece může mít za následek haploinsufficiency jednoho nebo více genů. Duplikace může vést k narušení exprese genu nebo může mít za následek haploinsufficiency genů, které jsou narušeny zlomy duplikace. Analýza rodičovských vzorků může být nezbytná pro správnou interpretaci výsledků.
Při prvním použití MLPA nebo pokud dojde ke změně metody izolace DNA nebo používaných přístrojů, je třeba provést interní validaci MLPA metody pomocí 16 vzorků DNA od zdravých jedinců. Při validaci by všechny sondy měly vykazovat standardní odchylku <0,10.
Neúplná denaturace DNA, v důsledku přítomnosti solí ve vzorku DNA, může vést k detekci falešně pozitivní delece vykazující snížený signál u všech sond, které se nacházejí v CpG ostrovech. Použití alternativní metody izolace DNA může řešit takové případy.
Falešně pozitivní duplikace, v důsledku kontaminace DNA vzorků cDNA nebo PCR amplikony jednotlivých exonů, byly také popsány (Varga et al, 2012). Analýza nezavisle odebraného sekundárního vzorku DNA může vyloučit vliv těchto kontaminačních artefaktů.
Normální počet kopií variant u zdravých jedinců je popsán v databázi genomových variant:
http://dgv.tcag.ca/dgv/app/home. Uživatelé by měli vždy ověřit nejnovější aktualizaci příslušných databází a seznámit se s vědeckou literaturou při interpretaci svých zjištění.
Databáze genomových variant a fenotypu: http://decipher.sanger.ac.uk/. Důrazně doporučujeme, aby uživatelé ukládali pozitivní výsledky v databázi Decipher. Nahlaste nám, prosím, změny v počtu kopií detekovaných u pacienta nebo rodičů, falešně pozitivní výsledky způsobené SNP a další neobvyklé výsledky (např. delece nebo duplikace zjištěné u několika nepříbuzných pacientů).
Bezpečnostní opatření a varování: Pouze pro profesionální použití. Před použitím se vždy podrobně seznamte s nejaktuálnějším popisem produktu a MLPA obecným protokolem: mlpa.com. Je odpovědností uživatele, aby bral na vědomí nejnovější vědecké poznatky o dané aplikace před vyslovením závěrů zjištěných tímto produktem.
Publikace uvádějící kit SALSA MLPA Probemix P036 Subtelomeres Mix 1
- Ahn J. et al. (2007). Detection of subtelomere imbalance using MLPA: validation, development of an analysis protocol, and application in a diagnostic centre. BMC Medical Genetics. 2007 Mar 5;8(1):9.
- Ahn J. et al. (2008). Submicroscopic chromosome imbalance in patients with developmental delay and/or dysmorphism referred specifically for Fragile X testing and karyotype analysis. Mol Cytogenet.1:2.
- Bendavid C. et al. (2007). MLPA screening reveals novel subtelomeric rearrangements in holoprosencephaly. Hum Mutat. 28:1189-97.
- Bruno D.L. et al. (2006). High-throughput analysis of chromosome abnormality in spontaneous miscarriage using anMLPA subtelomere assay with an ancillary FISH test for polyploidy. Am J Med Genet A. 140:2786-93.
- Christofolini DM. et al. (2010). Subtelomeric rearrangements and copy number variations in people with intellectual disabilities. J Intellect Disabil Res.54:938-42.
- Darnfors C et al. (2005). High-resolution analysis of the subtelomeric regions of human embryonic stem cells. Stem Cells.; 23:483-8.
- Delahaye A. et al. (2009). Chromosome 22q13.3 deletion syndrome with a de novo interstitial 22q13.3 cryptic deletion disrupting SHANK3. Eur J Med Genet. 52:328-32.
- Erjavec-Skerget A. et al. (2006). Subtelomeric chromosome rearrangements in children with idiopathic mental retardation: applicability of three molecular-cytogenetic methods. Croat Med J. 47:841-50.
- Faas BH. et al. (2008). Detection of cryptic subtelomeric imbalances in fetuses with ultrasound abnormalities. Eur J Med Genet. 51:511-9.
- Grati FR. et al. (2008). Prenatal detection by subtelomeric FISH and MLPA of unbalanced meiotic recombinants resulting from parental pericentric inversions. Mol.Cell Probes 22:316-9.
- Hila L. et al. (2009). MLPA subtelomere analysis in Tunisian mentally retarded patients. Biochem Genet. 47:727-33.
- Kirchhoff M. et al. (2005). Investigation of patients with mental retardation and dysmorphic features using comparative genomic hybridization and subtelomeric multiplex ligation dependent probe amplification. Am J Med Genet A. 139:231-3.
- Kjaergaard S. et al. (2010). Diagnostic yield by supplementing prenatal metaphase karyotyping with MLPA for microdeletion syndromes and subtelomere imbalances. Prenat Diagn. 30:995-9.
- Konialis C. et al. (2011). Uncovering recurrent microdeletion syndromes and subtelomeric deletions/duplications through non-selective application of a MLPA-based extended prenatal panel in routine prenatal diagnosis. Prenat Diagn. 31:571-7.
- Koolen DA et al. (2004). Screening for subtelomeric rearrangements in 210 patients with unexplained mental retardation using multiplex ligation dependent probe amplification (MLPA). J Med Genet.; 41:892-9.
- Lam AC et al. (2006) High rate of detection of subtelomeric aberration by using combined MLPA and subtelomeric FISH approach in patients with moderate to severe mental retardation. Clin Biochem. 39:196-202.
- Li MR. et al. (2009). Multiplex ligation-dependent probe amplification analysis of subtelomeric chromosome rearrangements in children with idiopathic mental retardation. Zhonghua Yi Xue Za Zhi 89:2839-42.
- Mademont-Soler I. et al. (2010). Subtelomeric MLPA: is it really useful in prenatal diagnosis? Prenat Diagn. 30:1165-9.
- Mandal K. et al. (2009). Use of Multiplex Ligation-Dependent Probe Amplification (MLPA) in screening of subtelomeric regions in children with idiopathic mental retardation. Indian J.Pediatr. 76:1027-31.
- Monfort S. et al. (2006). Evaluation of MLPA for the detection of cryptic subtelomeric rearrangements. J Lab Clin Med. 147:295-300.
- Mundhofir FE. et al. (2013). Subtelomeric chromosomal rearrangements in a large cohort of unexplained intellectually disabled individuals in Indonesia: A clinical and molecular study. Indian J Hum Genet. 19:171-8.
- Northrop, EL. et al. (2005). Detection of cryptic subtelomeric chromosome abnormalities and identification of anonymous chromatin using a quantitative multiplex ligation-dependent probe amplification (MLPA) assay. Hum Mutat. 26:477-86.
- Palomares, M. et al. (2006). MLPA vs multiprobe FISH: comparison of two methods for the screening of subtelomeric rearrangements in 50 patients with idiopathic mental retardation. Clin Genet. 69:228-33.
- Rooms, L. et al. (2004). Subtelomeric deletions detected in patients with idiopathic mental retardation using multiplex ligation-dependent probe amplification (MLPA). HumMutat. 23:17-21.
- Rooms, L. et al. (2005). Subtelomeric rearrangements in the mentally retarded: a comparison of detection methods. Hum Mutat. 25:513-24.
- Rooms, L. et al. (2006). Multiplex ligation-dependent probe amplification to detect subtelomeric rearrangements in routine diagnostics. Clin Genet. 69:58-64.
- Rosello M. et al. (2010). Prenatal study of common submicroscopic "genomic disorders" using MLPA with subtelomeric/microdeletion syndrome probe mixes among gestations with ultrasound abnormalities in the first trimester. Eur J.Med Genet. 53:76-9.
- Ruiter EM. et al. (2007). Pure subtelomeric microduplications as a cause of mental retardation. Clin.Genet.72:362-8.
- Rusu C. et al. (2009). Use of MLPA test in the detection of subtelomeric rearrangements--case report. Rev Med Chir Soc Med Nat Iasi. 113:516-22.
- Stegmann AP. et al. (2008). Prospective screening of patients with unexplained mental retardation using subtelomeric MLPA strongly increases the detection rate of cryptic unbalanced chromosomal rearrangements. Eur J Med Genet. 51:93-105.
- Stevens SJ. et al. (2009). Identical cryptic partial monosomy 20pter and trisomy 20qter in three adult siblings due to a large maternal pericentric inversion: detection by MLPA and breakpoint mapping by SNP array analysis. Am J Med Genet A. 149A:2226-30.
- Van Opstal D. et al. (2011). Multiplex ligation dependent probe amplification (MLPA) for rapid distinction between unique sequence positive and negative marker chromosomes in prenatal diagnosis. Mol.Cytogenet. 4:2.
- Verdu Perez A. et al. (2011). Subtelomeric rearrangements in cryptogenic mental retardation. An Pediatr.(Barc).75:365-71.
- Wu Y. et al. (2007). Detection of subtelomeric rearrangements in patients with idiopathic mental retardation/developmental delay. Zhonghua Er Ke Za Zhi. 45:906-11.
- Wu Y. et al. (2010). Submicroscopic subtelomeric aberrations in Chinese patients with unexplained developmental delay/mental retardation. BMC Med.Genet.11:72.
Další reference:
- Schouten J.P. et al. (2002). Relative quantification of 40 nucleic acid sequences by multiplex ligation-dependent probe amplification. Nucleic Acid Res. 30:e57 (Original description of the MLPA technique).
- Firth H.V. et al. (2009). DECIPHER: Database of Chromosomal Imbalance and Phenotype in Humans Using Ensembl Resources. Am J Hum Genet. 84:524-533
- Hills A. et al. (2010). MLPA for confirmation of array CGH results and determination of inheritance. Mol Cytogenet. 3:19.
- Jennings LJ. et al (2011). Validation of multiplex ligaton-dependent probe amplification for confirmation of array comparative genomic hybridization. Diagn Mol Pathol. 20:166-74.
- Shao L. et al. (2008). Identification of chromosome abnormalities in subtelomeric regions by microarray analysis: a study of 5,380 cases. Am J Med Genet A. 146A:2242-51.
- Van den Berg C. et al. (2006). (Potential) false-negative diagnoses in chorionic villi and a review of the literature.Prenat Diagn. 26:401-8.
- Varga R.E. et al. (2012) MLPA-based evidence for sequence gain: Pitfalls in confirmation and necessity for exclusion of false positives. Anal Biochem. 421:799-801.
Související kity SALSA MLPA
P070 Subtelomeres Mix 2A
Obsahuje jednu sondu pro každou ze 41 různých subtelomerických oblastí a 5 sond v blízkosti centromerické oblasti pěti akrocentrických chromozomů. Všechny sondy jsou odlišné od P036 sond. Důrazně doporučujeme používat oba kity P036 a P070 (nebo P069) u každého vzorku.
P069 Subtelomeres Mix 2B
Shodný s P070 ale neobsahuje sondy v blízkosti centromery akrocentrických chromozomů.
P245 Mikrodeletion-1
Sondy pro 23 různých mikrodelečních syndromů; lze použít pro primární skríning
mikrodelečních syndromů.
P064 MR-1
Sondy pro 15 různých mikrodelečních syndromů; lze použít pro primární skríning
mikrodelečních syndromů.
P106 MRX
Sondy pro několik genů podílejících se na X-vázané mentální retardaci.
P181 Centromere-1
Kit bsahuje jednu sondu pro každou ze 41 různých centromerických oblastí a další sondu v blízkosti centromerické oblasti všech pěti akrocentrických chromozomů, což znamená 2 sondy pro každý chromozom.
P182 Centromere-2
Podobně jako u kitu P181 centromere, ale všechny sondy jsou odlišné od P181 sond.
P147 1p36
Sondy pro 1p36 subtelomerické oblasti.
P208 telomere-6
Sondy pro 2p, 3p, 6p a 8p subtelomerické oblasti.
P358 telomere-15
Sondy pro 4p a 5p subtelomerické oblasti + 13qcen a 14qcen oblasti.
P365 telomere-14
Sondy pro 7p a 16p subtelomerické oblasti + 15qcen a 21qcen oblasti.
P230 telomere-17
Sondy pro 9p, 10p, 11p a 12p subtelomerické oblasti.
P249 telomere-8
Sondy pro 17p, 18p, 19p a 20p subtelomerické oblasti.
P264 telomere-9
Sondy pro 1q, 2q, 3q a 4q subtelomerické oblasti.
P277 telomere-10
Sondy pro 5q, 6q, 7q a 8q subtelomerické oblasti.
P286 telomere-11
Sondy pro 9q, 10q, 11q a 12q subtelomerické oblasti.
P291 telomere-12
Sondy pro 13q, 14q, 15q a 16q subtelomerické oblasti.
P320 telomere-13
Sondy pro 17q, 18q, 19q a 20q subtelomerické oblasti.
P356 chromosome 22q
Sondy pro 22q subtelomerické oblasti + oblast 22qcen.
+ 13, 14, 15, 21 a 22 q-cen sondy detekují q raménko v blízkosti centromery (akrocentrické chromozomy).
«Tato sonda je umístěna uvnitř nebo blízko ostrovu silně bohatého na CpG. Nízká úroveň signálu této sondy může být v důsledku neúplné denaturace vzorku DNA, například v důsledku přítomnosti solí ve vzorku DNA.
Důležité poznámky o specifických subtelomerách
DŮLEŽITÉ UPOZORNĚNÍ: několik sond je umístěno v oblastech, které vykazují variabilitu v počtu kopií u zdravých jedinců! Viz http://dgv.tcag.ca/dgv/app/home. Proto důrazně doporučujeme, vždy potvrdit výsledky vhodným navazujícím MLPA kitem nebo jinou metodou.
- 1p: P069/P070 sonda pro 1p detekuje sekvenci, která byla nalezena duplikovaná u normálních jedinců. Jelikož tato delece může mít nějaké projevy, nebudeme tuto sondu nahrazovat.
- 3p: Bylo zjištěno, že P036/P069/P070 sondy pro 3p (CHL1 gen) jsou duplikované u zdravých jedinců. Podle Dijkhuizen et al (2006;. Am J Hum Genet 140A :2482-87), defekty v
- centromerických genech CNTN4 a CRBN mohou být více důležité pro 3p syndrom než ztráta CHL1.
- 12p: The P036/P069/P070 sondy detekují sekvence, které jsou duplikované u zdravých jedinců.
- 13q-cen: Tato sonda detekuje sekvence na q-raménku v blízkosti centromery, zatímco p-raménko tohoto chromozomu obsahuje málo charakterizované geny.
- 14q-cen: Tato sonda detekuje sekvence na q-raménku v blízkosti centromery, zatímco p-raménko tohoto chromozomu obsahuje málo charakterizované geny.
- 15q-cen: Tato sonda detekuje sekvence na q-raménku v blízkosti centromery, zatímco p-raménko tohoto chromozomu obsahuje málo charakterizované geny. P036 sonda pro 15q-cen detekuje MKRN3 gen. Tato oblast je deletována u některých pacientů s Prader-Willi/Angelman syndromem. PWS / AS kritická oblast (SNRPN gen) se nachází ve vzdálenosti 1,3 Mb směrem k q-telomeře. Doporučujeme kit SALSA® probemix® MLPA ME028 Prader-Willi/Angelman pro další charakterizaci změn v počtu kopií regionu MKRN3.
- 16p: U několika zdravých jedinců byla zjištěna delece P036 sondy pro 16p, zatímco P070 sonda poskytovala normální výsledky.
- 20p: V P036/P069/P070 sondy pro 20p detekují sekvence, které jsou duplikovány ve sporadických případech u normálních jedinců. Jelikož tato delece může mít nějaké projevy, nebudeme tuto sondu nahrazovat.
- 21q-cen: Tato sonda detekuje sekvence na q-raménku v blízkosti centromery, zatímco p-raménko tohoto chromozomu obsahuje málo charakterizované geny.
- 22q-cen: Tato sonda detekuje sekvence na q-raménku v blízkosti centromery, zatímco p-raménko tohoto chromozomu obsahuje málo charakterizované geny.
- 3q: Bylo zjištěno, že P036 sonda pro 3q (BDH1 gen) je duplikována i u zdravých rodičů ve studii E. Reyniers, Antverpy (potvrzeno FISH). Sonda P070 / P069 pro 3q se nachází 200 kb blíže k telomeře a tuto duplikaci nevykazuje. Zdá se, že polymorfismus v počtu kopií genu BDH1 se vyskytuje u zdravých jedinců. Uvědomte si však, že 3q29 mikrodeleční syndrom (v důsledku intersticiální delece) byl popsán (Willatt et al, J. Hum Genet 77:154-160, 2005), a že sonda v kitu P036 detekuje sekvenci v tomto běžně deletovaném regionu. 3q sonda v kitu P070 nezjistí tuto mikrodeleci, neboť detekuje sekvenci umístěnou mezi intersticiální delecí a telomerou.
- 4q: 4q telomerická oblast je složitá a obsahuje velmi málo genů. Pouze gen FRG1 je dobře charakterizován na konci 2-3 Mb 4q, ale většina testovaných specifických sond pro FRG1 vykazovala nespolehlivé výsledky v důsledku přítomnosti populačně specifických SNP v genu FRG1. 4q sonda P036-E se nachází ve větší vzdálenosti od telomery; neočekáváme, že by to vedlo k falešně negativním výsledkům, navíc delece telomerické oblasti 2-3Mb 4q zřejmě nemá žádné fenotypové projevy (2008; Shao, L.et al. Am J Med Genet. 146A: 2242-51).
- 5q: Bylo zjištěno u jednoho pacienta, že sonda pro P036 5q byla ovlivněna polymorfismem v prvním nukleotidu za ligačním místem. K P036 / P069 / P070 sondy pro 5q detekují sekvence, které jsou duplikovány ve sporadických případech u normálních jedinců. Jelikož delece této oblasti může mít nějaké projevy, nebudeme tuto sondu nahrazovat.
- 12q: Byla nalezena ZNF10 duplikace u zdravého rodiče detekovaná P036 i P070.
- 19q: Sonda P036 / P070 pro 19q dala různé výsledky u zdravých kontrol.
- 22q: Bylo zjištěno, že P070 sonda pro 22q byla deletována u jednoho zdravého jedince. Zjištěná sekvence pomocí sondy P036 pro 22q obsahuje pouze jeden mismatch v porovnání s příbuznou sekvencí na chromozomu 22. To je však dostatečné pro generování chr. 22q specifického signálu při MLPA. Nicméně když tato oblast má být sekvenována, návrh primerů je složitý, neboť 4 mismatch jsou přítomny ve 350 nt oblasti obsahující tuto cílovou sekvenci sondi. Kit P188 obsahuje mnoho sond v blízkosti 22q13 telomery, které mohou být použity pro validaci nebo pro další charakterizaci 22q13 delece.
- X, Y: Sondy specifické pro pohlavní chromozomy v kitech P036/P069/P070 detekují sekvence umístěné na obou X a Y chromozomech v blízkosti telomer (PAR region) a budou tak indikovat kombinaci počtu kopií X a Y.
Hledaní genetické příčiny vrozených vad pomocí metody MLPA
Počet genů, jejichž defekt může mít za následek opožděný vývoj, je velký. V některých případech, zejména fenotypové znaky indikují zapojení určitého genu nebo chromozomální oblasti. Mnoho SALSA MLPA kitů je k dispozici pro detekci příčin opožděného vývoje s výraznými syndromovými rysy, jako jsou syndromy RETT, Sotos a Prader-Willi/Angelmanův.
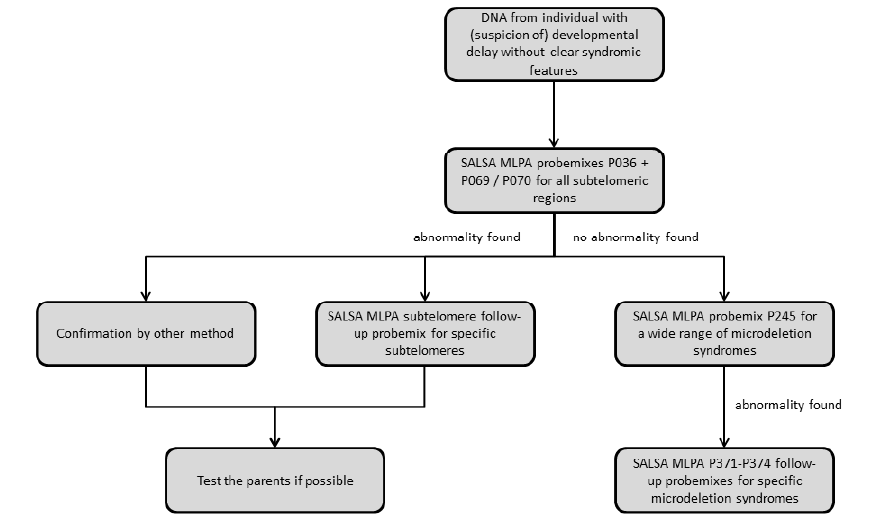
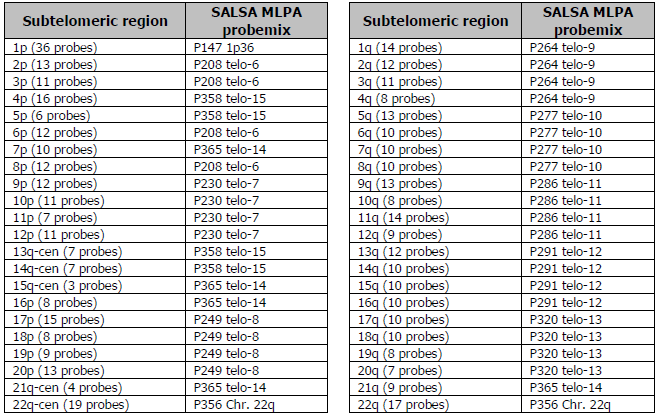
U jedinců (s podezřením) s vrozenými abnormalitami je genetická příčina známa pouze v menšině případů. Používáte-li kity P036 a P069/P070, doporučujeme postupovat podle strategie uvedené v Obrázku 1. Tyto kity mohou být použity pro široký skríning počtu kopií subtelomerických variant. V případě, že pomocí kitů P036/P069/P070 detekujete nějakou abnormalitu, tento výsledek by měl být potvrzen buď některým z navazujících MLPA kitů (viz tabulka níže) nebo jinou metodou (např. FISH, array-CGH). Navazující
kity pro validace obsahují více sond pro subtelomery a poskytují rychlý způsob, jak přezkoumat určité oblasti podrobněji. Navazující MLPA kity Vám umožní potvrdit přítomnost aberace a odhadnout její velikost, ale budou poskytovat méně informací ve srovnání s array CGH a sekvenováním genomu s nízkým pokrytím.
V některých případech je vhodné provést skríning DNA od rodičů, pokud jsou k dispozici. V případě, že není nalezena žádná abnormalita, je možné použít SALSA MLPA kit P245 pro skríning různých mikrodelečních syndromů. Je-li test pozitivní, výsledky mohou být dále charakterizovány navazujícími kity P371-P374 pro specifické syndromy. Podrobnější popis různých kitů naleznete níže.
Obrázek 1. Doporučený postup pro testování jedinců s podezřením na opožděný vývoj bez jasných syndromových znaků.
Kity SALSA® MLPA® probemix P036 Subtelomeres Mix 1 a P069 nebo P070 Subtelomeres
Při správném použití, skríning pomocí MLPA kitů P036 a P069 / P070 vyloučí přítomnost
abnormálního počtu kopií subtelomerických regionů ve většině vzorků. MLPA však nedetekuje inverze nebo balancované translokace. Studie ukázaly, že 3 - 8% všech případů s opožděným vývojem je způsobeno abnormálním počtem kopií subtelomerických oblastí (viz odkazy na publikace). SALSA® MLPA® kit P036 Subtelomeres Mix 1 a P070 Subtelomeres Mix 2B obsahují jednu sondu pro každou subtelomeru a mohou být použity pro detekci většiny delecí / duplikací v subtelomerických oblastech. Velmi doporučujeme používat oba kity společně s cílem maximalizovat záchyt aberací.
Většina sond v kitech P036 a P070 je zaměřena na dobře charakterizované geny v krátké vzdálenosti v blízkosti telomer. Výjimka je stanovena pro p-raménka chromozomů 13, 14, 15, 21 a 22, neboť tyto chromozomy obsahují více než 10 Mb repetitivních sekvencí. Tyto "p" sondy rozpoznávají jeden z prvních genů na q- raménku v blízkosti centromery. SALSA® MLPA® kit P069 Subtelomeres Mix 2A neobsahuje sondy pro tyto akrocentrická raménka.
Interpretace abnormálního počtu kopií v subtelomerických oblastech je složitá. Změny v počtu subtelomerických kopií se mohou také vyskytovat u zdravých jedinců a efekt delece nebo duplikace závisí na postiženém genu. Značný počet abnormalit detekováných jedinou sondou nemusí být příčinou jakéhokoliv fenotypového projevu, ale může to být v důsledku vzácného polymorfismu nebo změny v počtu kopií, která je rovněž přítomna u jednoho z rodičů. U některých chromozomálních ramének, i velké subtelomerické delece nebo duplikace (> 1 MB) mohou být zděděny bez jasného fenotypového projevu; viz poznámky níže. Ne všechny abnormality detekované pomocí kitů P036 a P069 / P070 jsou patogenní. U všech detekováných abnormalit, doporučujeme testování rodičů a zjistit, zda aberantní počet kopií vznikl u pacienta de novo. De novo delece / duplikace mají vyšší pravděpodobnost, že budou příčinou opožděného vývoje. Databáze genomových variant může být užitečná pro ověření, zda ke změnám v počtu kopií dochází i u normálních jedinců: http://projects.tcag.ca/variation/.
Podrobnější analýza subtelomerických oblastí Pro bližší zkoumání vzorků, u kterých byly nalezeny abnormální počty kopií, jsou k dispozici další MLPA kity.
Kit P245 Microdeletion -1 obsahuje sondy pro 23 různých mikrodelečních syndromů, které způsobují opožděný vývoj. V případě nalezení abnormality pomocí tohoto kitu, doporučujeme další vyšetření pomocí jednoho z mikrodelečních navazujících kitů. Viz detailní informace v manuálu pro kit P245 Microdeletion-1.